F: Tratamiento de una imagen científica (Ha-R)¶
Vamos a estudiar las regiones HII de la galaxia NGC6118. Para ello tendremos que preparar dos imágenes con diferentes filtros, Ha y R ya que sabemos que el filtro Ha es un filtro estrecho centrado en la banda 6607A y el filtro R, aunque está centrado en esas longitudes de onda, es más amplio.
Por lo tanto, lo que haremos es realizar una fotometría de las estrellas en la imagen Ha ya que tiene menos estrellas que la imagen R y a partir de la imagen R podemos añadir alguna estrella que no esté en Ha.
Una vez realizada la fotometría, debemos graficar los flujos de los dos filtros y trazar la recta de ajuste. Así conseguiremos realizar una estimación de la diferencia de flujo debido a los filtros.
Localizar estas regiones es importante en astronomía porque nos pueden ayudar a estudiar la velocidad radial de las galaxias como proponen Kam, Z. S et. al.
Abriendo las imágenes¶
[1]:
import matplotlib.pyplot as plt
import numpy as np
from astropy.io import fits
import os
import pandas as pd
import matplotlib.patches as patches
import scipy
from photutils import aperture_photometry
from photutils import CircularAperture
from astropy.stats import sigma_clipped_stats
from photutils import DAOStarFinder
from photutils import make_source_mask
from mpl_toolkits.mplot3d import Axes3D
from photutils.isophote import EllipseGeometry
from sklearn.linear_model import LinearRegression
from sklearn import linear_model
from sklearn.metrics import mean_squared_error, r2_score
[2]:
ha=fits.open("imagenes/regionHII/ha.fit")[0].data[400:-400,400:-400]
r=fits.open("imagenes/regionHII/r.fit")[0].data[400:-400,400:-400]
Detectando estrellas en Ha¶
DAOStarFinder Stetson 1987; PASP 99, 191 busca en las imágenes los máximos de densidad en el flujo.
[3]:
mean, median, std = sigma_clipped_stats(ha, sigma=3.0) # definimos una mediana con sigma_clipped_stats
daofind = DAOStarFinder(fwhm=3.0, threshold=80.*std) # detectamos las fuentes con DAOStarFinder
mask = np.zeros(ha.shape, dtype=bool) # Generamos una mascara para qutar el centro de la imagen
mask[200:1000, 200:1000] = True
sources = daofind(ha - median, mask=mask) # detectamos las fuentes que esten por encima de la mediana
for col in sources.colnames:
sources[col].info.format = '%.8g' # formateo de la tabla
print(sources)
id xcentroid ycentroid sharpness ... sky peak flux mag
--- --------- --------- ---------- ... --- ---------- --------- ------------
1 349.08407 23.06115 0.45760264 ... 0 0.28782383 1.120405 -0.12343756
2 1252.3425 111.83957 0.49992619 ... 0 1.7884092 7.9329739 -2.2485901
3 100.84967 141.65812 0.4519161 ... 0 2.009676 7.3235307 -2.1618013
4 139.75055 154.01524 0.49915704 ... 0 0.39054054 1.4219631 -0.38222081
5 398.0879 182.65515 0.47621736 ... 0 0.36278591 1.5117347 -0.44868897
6 1242.7372 644.93147 0.41871981 ... 0 0.51455635 2.4777501 -0.98514374
7 1050.4904 648.32583 0.51884046 ... 0 0.64771599 2.9557924 -1.1766848
8 565.11216 1021.5083 0.4412611 ... 0 0.30307603 1.3398254 -0.31762051
9 299.00671 1132.516 0.46488908 ... 0 0.49880373 2.1088767 -0.81012797
10 327.52825 1144.4042 0.42840471 ... 0 11.307342 47.39386 -4.1893052
11 245.92845 1271.7488 0.45522224 ... 0 0.41422138 1.7048297 -0.5792025
12 660.31653 1274.4656 0.49179181 ... 0 0.25255319 1.0765691 -0.080104756
[4]:
plt.figure("Stars in Ha", figsize=[10, 10])
positions = np.transpose((sources['xcentroid'], sources['ycentroid']))
apertures = CircularAperture(positions, r=10.)
plt.imshow(ha,vmin=0, vmax=0.01, origin='lower')
apertures.plot(color='red', lw=1.5, alpha=0.5)
plt.show()

Una vez detectadas las fuentes calculamos la apertura circular en un radio de 20 píxeles:
[5]:
aperture = CircularAperture(positions, r=20.)
Y calculamos la fotometría de dicha apertura:
[6]:
phot_table_Ha = aperture_photometry(ha, aperture)
phot_table_Ha
[6]:
| id | xcenter | ycenter | aperture_sum |
|---|---|---|---|
| pix | pix | ||
| int64 | float64 | float64 | float64 |
| 1 | 349.0840718721037 | 23.06115023990617 | 5.9707853144109375 |
| 2 | 1252.3425282476803 | 111.8395660192342 | 31.12198779628524 |
| 3 | 100.84967175575383 | 141.65812290255334 | 46.177683387858465 |
| 4 | 139.75055253162196 | 154.01523501983976 | 9.187212283913759 |
| 5 | 398.0878982067919 | 182.65514850657365 | 7.112305744900143 |
| 6 | 1242.7372495712386 | 644.9314689138399 | 8.795779939807034 |
| 7 | 1050.4903937456747 | 648.3258263525412 | 10.492986704029342 |
| 8 | 565.1121596416924 | 1021.5082530499211 | 5.3790705228001165 |
| 9 | 299.00671132484865 | 1132.5160282322252 | 11.273667917231116 |
| 10 | 327.52824953169124 | 1144.4041834299107 | 227.57507170675046 |
| 11 | 245.92844626442664 | 1271.7487790690082 | 8.20190432025236 |
| 12 | 660.3165326153387 | 1274.4656046219322 | 4.054421322970487 |
[7]:
plt.figure("Stars in R", figsize=[10, 10])
positions = np.transpose((sources['xcentroid'], sources['ycentroid']))
apertures = CircularAperture(positions, r=10.)
plt.imshow(r, vmin=np.nanmin(r), vmax=np.nanmedian(r)*800, origin='lower')
apertures.plot(color='red', lw=1.5, alpha=0.5)
plt.show()

[8]:
phot_table_R = aperture_photometry(r, aperture)
phot_table_R
[8]:
| id | xcenter | ycenter | aperture_sum |
|---|---|---|---|
| pix | pix | ||
| int64 | float64 | float64 | float64 |
| 1 | 349.0840718721037 | 23.06115023990617 | 198.1431176575303 |
| 2 | 1252.3425282476803 | 111.8395660192342 | 1236.7766704090466 |
| 3 | 100.84967175575383 | 141.65812290255334 | 1690.1940084358525 |
| 4 | 139.75055253162196 | 154.01523501983976 | 307.37253223122707 |
| 5 | 398.0878982067919 | 182.65514850657365 | 231.46341415609555 |
| 6 | 1242.7372495712386 | 644.9314689138399 | 334.2297095809117 |
| 7 | 1050.4903937456747 | 648.3258263525412 | 360.05377588415286 |
| 8 | 565.1121596416924 | 1021.5082530499211 | 197.14452375154906 |
| 9 | 299.00671132484865 | 1132.5160282322252 | 451.45046283292936 |
| 10 | 327.52824953169124 | 1144.4041834299107 | 8390.575172524223 |
| 11 | 245.92844626442664 | 1271.7487790690082 | 296.45947593985693 |
| 12 | 660.3165326153387 | 1274.4656046219322 | 154.4934309550923 |
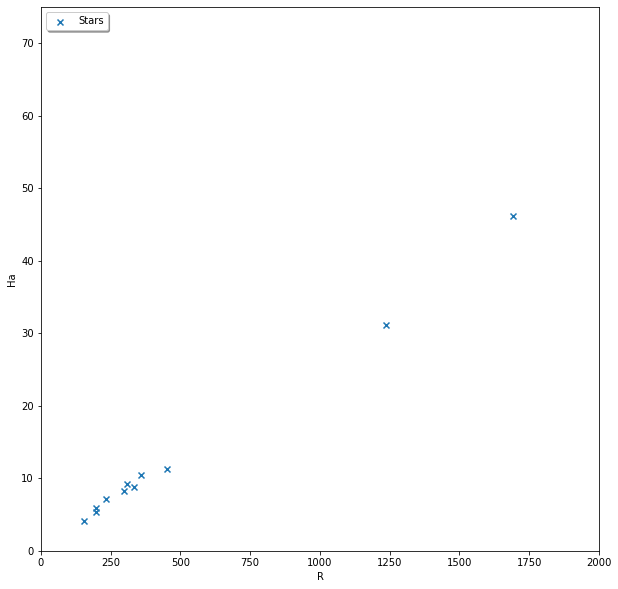
Ha vs R¶
Comparamos el flujo de el filtro de H-alpha y el del canal rojo:
[9]:
plt.figure("Ha vs R", figsize=[10, 10])
plt.scatter(phot_table_R['aperture_sum'],phot_table_Ha['aperture_sum'] , marker='x', alpha=1, label='Stars')
plt.xlim(0, 2000)
plt.ylim(0, 75)
plt.legend (loc='upper left', shadow=True, fontsize='medium')
plt.ylabel("Ha")
plt.xlabel("R")
plt.show()
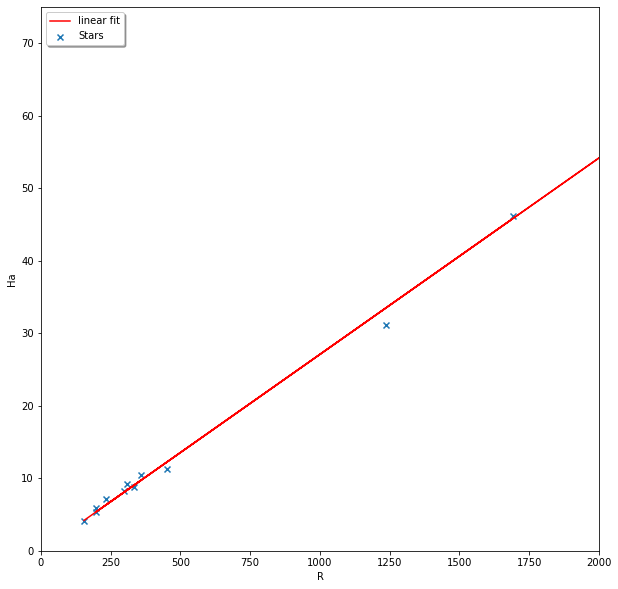
Ajuste lineal: \(Ha = aR+b\)¶
Realizamos un ajuste lineal para determinar la compensación de flujo:
[10]:
# y = ax + b
regresion_lineal = LinearRegression()
regresion_lineal.fit(np.asarray(phot_table_R['aperture_sum']).reshape(-1,1), np.asarray(phot_table_Ha['aperture_sum']))
a = regresion_lineal.coef_
b = regresion_lineal.intercept_
print('a = ' + str(a) + ', b = ' + str(b))
a = [0.02709269], b = 0.012804110295842719
\(R^2\)¶
[11]:
r2 = regresion_lineal.score(np.asarray(phot_table_R['aperture_sum']).reshape(-1,1), np.asarray(phot_table_Ha['aperture_sum']))
print('Coeficiente de determinacion R2 = ' + str(r2))
Coeficiente de determinacion R2 = 0.999787706607886
Graficando Ha vs R y su ajuste lineal¶
[12]:
plt.figure("Ha vs R", figsize=[10, 10])
y_ajuste = phot_table_R['aperture_sum']*a + b
# Dibujamos la recta de ajuste
plt.plot(phot_table_R['aperture_sum'], y_ajuste, 'r-', label ='linear fit')
plt.scatter(phot_table_R['aperture_sum'],phot_table_Ha['aperture_sum'] , marker='x', alpha=1, label='Stars')
plt.xlim(0, 2000)
plt.ylim(0, 75)
plt.legend (loc='upper left', shadow=True, fontsize='medium')
plt.ylabel("Ha")
plt.xlabel("R")
plt.show()
Multiplicando la imagen del filtro R por a¶
[13]:
r_scale = r * a
Sustrayendo la imagen R de la imagen Ha¶
[14]:
Ha_R = ha - r_scale
[15]:
Ha_R
[15]:
array([[ 5.87614958e-04, -2.22705754e-03, -6.33193890e-05, ...,
-1.95511944e-03, -2.19005413e-03, -1.07355891e-03],
[ 1.44865865e-03, 1.68791693e-03, -2.92267763e-03, ...,
-5.02979401e-03, -1.32460991e-04, 2.46133466e-03],
[-1.45900960e-03, -1.07090869e-03, 6.85737659e-04, ...,
-1.65304801e-03, -2.56703439e-03, 3.42935420e-04],
...,
[-2.10557561e-03, 3.17371688e-03, -1.01978432e-03, ...,
-8.15871924e-03, -9.66137502e-03, -6.33899882e-03],
[-3.50515216e-04, -1.37433936e-04, 3.51346817e-03, ...,
-9.14807545e-03, -4.38527888e-03, -9.54893225e-03],
[-1.00476876e-03, 1.81116940e-03, 1.69438158e-03, ...,
-5.03344479e-03, -8.53135309e-03, -6.68820593e-03]])
Graficando Ha-R¶
[16]:
plt.figure("Ha-R", figsize=[10, 10])
plt.imshow(Ha_R, vmin=0, vmax=0.01, origin='lower')
plt.colorbar(label='Counts')
plt.show()

Guardando Ha-R¶
[17]:
hdu_Ha = fits.PrimaryHDU(Ha_R.astype(np.float32))
hdu_Ha.writeto('salidas/Ha_R.fit', overwrite=True)
Buscando las regiones HII en nuestra imagen¶
La galaxia no está de frente: tiene un ángulo de inclinación que debemos corregir. Para ello primero rotaremos la imagen y luego aplicaremos una transformación a los ejes (tenemos una galaxia con forma de elipse y sabemos que las galaxias espirales son circulares).
Utilizaremos el paquete ndimage de SciPy.
Rotando la imagen¶
[18]:
from scipy import ndimage, misc
[19]:
#geometry = EllipseGeometry(x0=658, y0=682, sma=20, eps=0.5,pa=20.*np.pi/180.)
[20]:
Ha_R_rotate = scipy.ndimage.rotate(Ha_R, 32, reshape=False, output=None, order=3, mode='constant', cval=0.0, prefilter=True)
[21]:
plt.figure("master_Ha-R scale", figsize=[10, 10])
plt.imshow(Ha_R_rotate, vmin=0, vmax=0.01, origin='lower')
plt.colorbar(label='Counts')
plt.show()
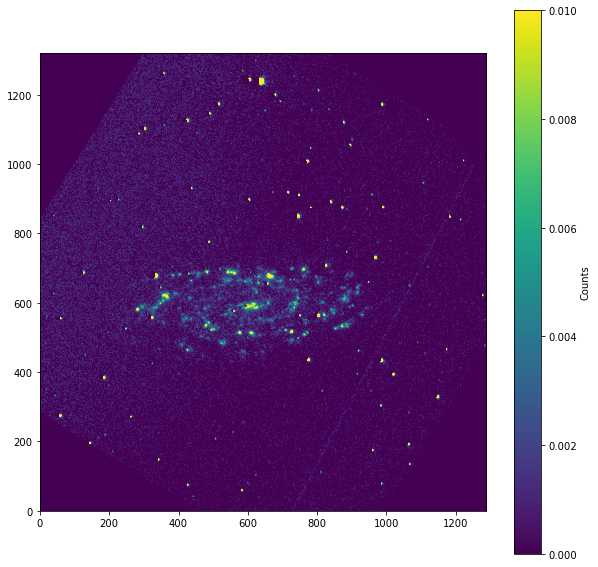
Transformando la galaxia eliptica en galaxia circular¶
[22]:
transform = ndimage.zoom(Ha_R_rotate, [3, 1])
[23]:
plt.figure("master_Ha-R scale", figsize=[10, 10])
plt.imshow(transform[1200:2300,100:1100], vmin=0, vmax=0.01, origin='lower')
plt.colorbar(label='Counts')
plt.show()
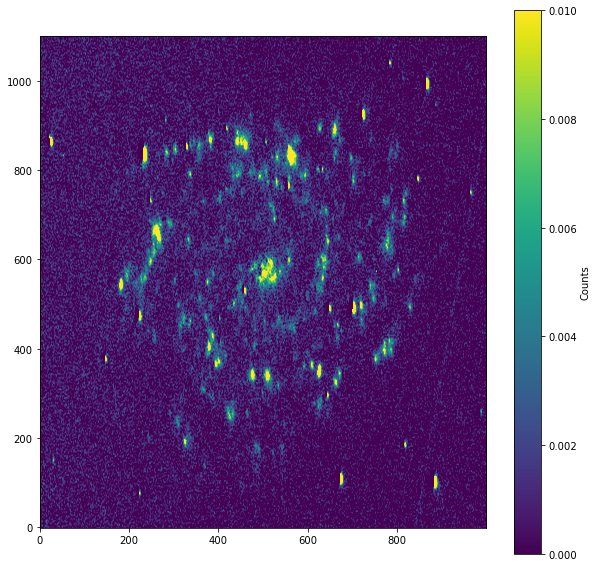
Buscando las regiones de Ha¶
Ahora vamos a buscar las regiones HII de nuestra galaxia.
Para ello usaremos la herramienta que habíamos utilizado anteriormente para la detección de estrellas DAOStarFinder.
Encontramos 113 fuentes de HII aunque tenemos que tener cuidado con los parámetros que elegimos ya que se nos puede colar estructura que no queremos o estrellas. Para evitar esto, es bueno utilizar máscaras para evitar regiones fuera de la galaxia.
[24]:
mean_H, median_H, std_H = sigma_clipped_stats(transform[1200:2300,100:1100], sigma=0.8)
daofind_H = DAOStarFinder(fwhm=10, threshold=150*std_H, sharplo = 0., sharphi=200, roundlo=0.01, roundhi=500)
star_mask = np.zeros(transform[1200:2300,100:1100].shape, dtype=bool)
star_mask[:200,0:250] = True
star_mask[:,0:125] = True
star_mask[879:,:] = True
star_mask[:,835:] = True
star_mask[0:226,674:] = True
star_mask[820:,0:247] = True
sources_H = daofind_H(transform[1200:2300,100:1100] - median_H, mask=star_mask)
for col in sources_H.colnames:
sources_H[col].info.format = '%.8g' # for consistent table output
print(sources_H)
id xcentroid ycentroid sharpness ... sky peak flux mag
--- --------- --------- ------------ ... --- ------------ --------- ------------
1 673.34791 108.28638 0.58854268 ... 0 0.28304639 89.848598 -4.8837783
2 462.70304 258.05397 0.54279086 ... 0 0.0046983214 1.2345386 -0.22876165
3 624.21743 274.30205 0.60176744 ... 0 0.0064568319 1.0659275 -0.069319183
4 613.55167 278.6011 0.43907255 ... 0 0.003923736 1.0261012 -0.027975484
5 643.86824 296.38696 0.63987169 ... 0 0.014474816 3.5996975 -1.390665
6 632.08163 300.79437 0.31789377 ... 0 0.0013197177 1.1323417 -0.13494377
7 532.54896 318.6302 0.81185107 ... 0 0.0067532225 1.1485891 -0.15041173
8 508.69781 339.63691 0.57408677 ... 0 0.02770378 4.9978188 -1.7469513
9 669.65575 345.57069 0.58891515 ... 0 0.011462141 2.5940775 -1.0349574
10 624.1418 349.24394 0.64078115 ... 0 0.050128334 10.201395 -2.5216489
... ... ... ... ... ... ... ... ...
101 381.30952 853.14788 0.78079617 ... 0 0.0070736353 1.1861123 -0.1853145
102 328.84897 851.59042 0.34346848 ... 0 0.016070412 7.2664881 -2.1533114
103 355.89246 855.33059 0.34203403 ... 0 0.0067478149 1.6131511 -0.51918761
104 458.67073 856.32579 0.43982402 ... 0 0.019264253 3.3750725 -1.3207078
105 450.24039 862.20012 0.8537384 ... 0 0.015236503 1.4662118 -0.41549181
106 506.03888 862.30651 0.57528107 ... 0 0.0091484959 3.1322564 -1.2396433
107 440.66565 864.09099 0.56855257 ... 0 0.017679877 3.0222884 -1.2008398
108 670.41467 867.16907 0.55048246 ... 0 0.005257102 1.1646337 -0.16547335
109 739.44706 868.26639 0.21047435 ... 0 0.0019689948 1.0423443 -0.045028041
110 380.66416 868.77978 0.69323155 ... 0 0.027843254 5.4233501 -1.8356691
111 656.68877 873.95501 0.0093337942 ... 0 0.0033410237 1.060428 -0.063703009
Length = 111 rows
[25]:
plt.figure("HII regions", figsize=[10, 10])
positions_H = np.transpose((sources_H['xcentroid'], sources_H['ycentroid']))
apertures_H = CircularAperture(positions_H, r=10.)
plt.imshow(transform[1200:2300,100:1100], vmin=0, vmax=0.01, origin='lower')
plt.colorbar(label='Counts')
apertures_H.plot(color='red', lw=1.5, alpha=0.6)
plt.show()
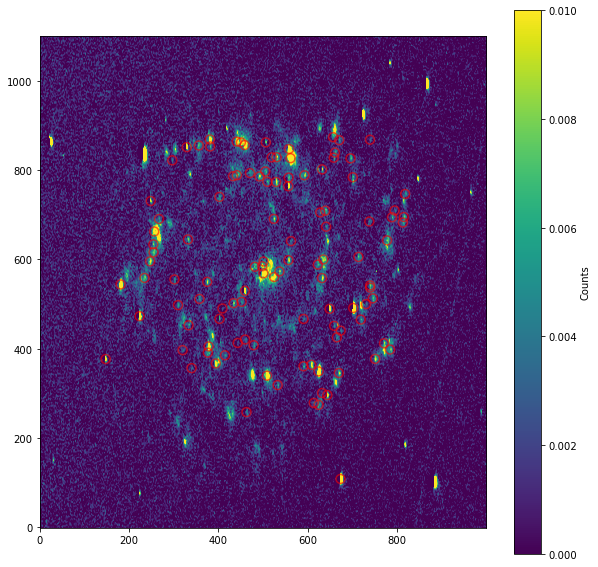
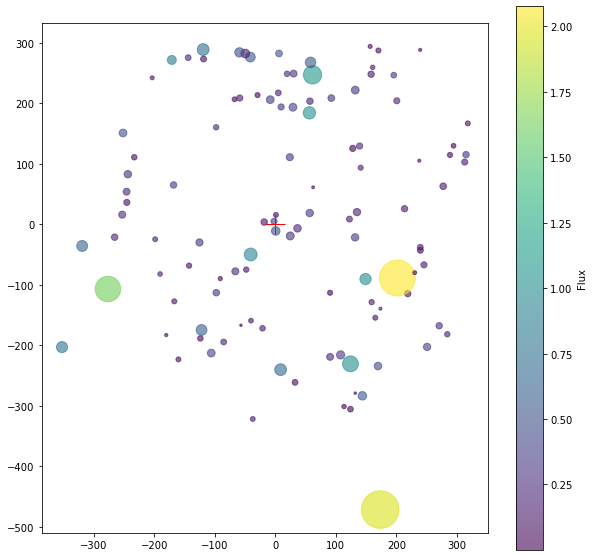
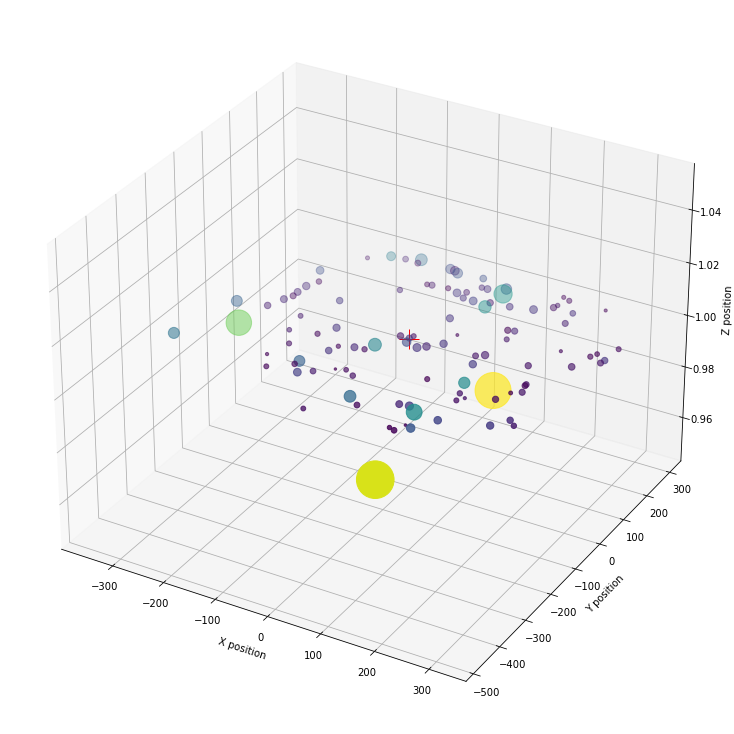
Trazando todas las fuentes de HII encontradas en la galaxia¶
Ahora vamos a representar estas regiones en 2D y 3D.
Las posiciones que nos ha ofrecido daofind son respecto a la imagen, por lo tanto vamos a recalcular todas las posiciones respecto al centro de la galaxia.
En el gráfico hemos rescalado el flujo que nos da la barra de color para tener un mayor contraste (aplicando el logaritmo a los datos).
También hemos reescalado el tamaño de las regiones HII, hemos tomado el pico de la distribución medido por daofind y lo hemos multiplicado por 5000.
Para hacer la representación en 3D hemos supuesto que la anchura de la galaxia es muy pequeña comparada con el tamaño de los brazos.
Centro de la galaxia¶
[26]:
H_x_center = 0 #500
H_y_center = 0 #580
Centrando los valores del eje x¶
[27]:
H_x = sources_H['xcentroid'] -500
Centrando los valores del eje y¶
[28]:
H_y = sources_H['ycentroid'] -580
Gráfico de dispersión (Sccater plot)¶
[29]:
plt.figure('Distribution of HII regions in 2D', figsize=[10, 10])
plt.subplot(111, aspect = 'equal')
plt.scatter(H_x, H_y, vmin=np.log10(np.min(sources_H['flux']))/2,
vmax=np.log10(np.max(sources_H['flux'])), c=np.log10(sources_H['flux']),
s=5000*sources_H['peak'], alpha=.6)
plt.colorbar(label='Flux')
plt.plot(H_x_center, H_y_center, 'r' ,marker='+', alpha=1, markersize=20)
[29]:
[<matplotlib.lines.Line2D at 0x7f894c5c0bb0>]
[30]:
fig = plt.figure('Distribution of HII regions in 3D', figsize=[10, 10])
ax = Axes3D(fig, auto_add_to_figure=False)
fig.add_axes(ax)
ax.scatter(H_x, H_y, 1 , vmin=np.log10(np.min(sources_H['flux'])),
vmax=np.log10(np.max(sources_H['flux'])),
c=np.log10(sources_H['flux']),
s=5000*sources_H['peak'])
ax.set_xlabel('X position')
ax.set_ylabel('Y position')
ax.set_zlabel('Z position')
plt.plot(H_x_center, H_y_center,1, 'r' ,marker='+', alpha=1, markersize=20)
plt.show()
Exportación de datos para una simulación de la rotación de la galaxia.¶
Podemos exportar los datos para poder hacer una simulación muy básica de la rotación de la galaxia.
[31]:
rotate = ({"x":H_x, "y":H_y, "flux":sources_H['flux'], "peak":sources_H['peak']})
[32]:
table_df = pd.DataFrame(rotate)
table_df.to_csv('salidas/table_rotation.csv')