E: Ejemplo real de calibración. Primera fase: bias¶
En este y los siguientes notebooks vamos a trabajar apilando y calibrando imágenes reales (los ficheros de las mismas deben encontrarse en la carpeta imagenes.
[1]:
import matplotlib.pyplot as plt
import numpy as np
import glob
from astropy.io import fits
import os
Esta es una función que «simplemente» muestra algunas estadísticas sobre una imagen.
[2]:
def imstats(a):
""""
Returns the statistics of an array.
Parameters
----------
a: array_like
Imput data
Returns
-------
d : dictionary
Number of elements, minimun, maximun, mean, median, standard
Examples
--------
>>> a = np.array([[1, 2], [3, 4]])
>>> imstats(a)
"""
x, y = np.shape(a)[0], np.shape(a)[1]
d = {'npix': x*y, 'min': np.nanmin(a), 'max': np.nanmax(a), 'mean': np.nanmean(a),'median': np.nanmedian(a), 'std': np.nanstd(a)}
return d
Como vamos a trabajar con varios ficheros intermedios es recomendable crear un nuevo directorio donde guardaremos las imágenes que se producirán en la calibración. Para ello usaremos la biblioteca os y el método .mkdir(): crearemos este directorio con un nombre concreto en mi caso lo he llamado imagenes/salidaImagenes.
[3]:
directorioCalibracion = 'salidas/salidaCalibracion/'
try:
os.mkdir(directorioCalibracion)
except:
print(f"El directorio {directorioCalibracion} ya existe")
Apertura de los Bias¶
Podemos abrir distintos archivos a la vez que se encuentren en un directorio dado. En este caso estamos dirigiéndonos al directorio ìmagenes/bias y estamos viendo los archivos que se encuentran dentro de este directorio con una extensión .fit, es decir el formato de la imagen.
Para ver que los archivos que tenemos en un directorio concreto podemos hacer un ls desde una celda concretando el directorio que queremos mostrar. Para sistemas operativos distintos a Windows las barras de los directorios son / y para Windows son \
[4]:
ls imagenes/calibracionImagenes/bias/*.fit #Para Linux / MAC
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0534_000001060.fit
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0535_000001061.fit
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0535_000001062.fit
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0536_000001063.fit
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0536_000001064.fit
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0536_000001065.fit
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0537_000001066.fit
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0537_000001067.fit
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0538_000001068.fit
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0538_000001069.fit
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0547_000001089.fit
[5]:
#ls imagenes\calibracionImagenes\bias\*.fit # Para Windows
Para poder listar tanto la ruta del archivo como el propio nombre del archivo lo podemos hacer de varios métodos. Aquí usamos la biblioteca glob que permite obtener la ruta de los archivos que se encuentren dentro del directorio indicado.
[6]:
bias_list = sorted(glob.glob('imagenes/calibracionImagenes/bias/*.fit'))
bias_list
[6]:
['imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0534_000001060.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0535_000001061.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0535_000001062.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0536_000001063.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0536_000001064.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0536_000001065.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0537_000001066.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0537_000001067.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0538_000001068.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0538_000001069.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0547_000001089.fit']
Una vez que tenemos nuestra lista de archivos bias, procedemos a abrir los archivos FITS que se encuentran en nuestra lista a través de la biblioteca AstroPy usando el método .open() del objeto fits.
Estas archivos los guardaremos en una nueva lista llamada hdul (recuerdemos: header data unit list) en la que tendremos tanto los metadatos (header) de las imágenes como los propios datos de la imagen (data).
[7]:
bias_data = []
bias_header = []
for bias in bias_list:
hdul_bias = fits.open(bias)
bias_data.append(hdul_bias[0].data)
bias_header.append(hdul_bias[0].header)
hdul_bias.close()
hdul_bias.info() # Mostramos la información de la primera imagen
Filename: imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0547_000001089.fit
No. Name Ver Type Cards Dimensions Format
0 PRIMARY 1 PrimaryHDU 45 (4096, 4096) uint16
Nos podemos asegurar que las imágenes que hayamos abierto son bias accediendo a los metadatos y viendo el tipo de imagen que tenemos. Esta clave en la cabecera la genera el propio programa de captura de imágenes y deberemos conocer el nombre con el que se ha designado.
[8]:
bias_header[0]
[8]:
SIMPLE = T
BITPIX = 16 /8 unsigned int, 16 & 32 int, -32 & -64 real
NAXIS = 2 /number of axes
NAXIS1 = 4096 /fastest changing axis
NAXIS2 = 4096 /next to fastest changing axis
BSCALE = 1.0000000000000000 /physical = BZERO + BSCALE*array_value
BZERO = 32768.000000000000 /physical = BZERO + BSCALE*array_value
DATE-OBS= '2020-01-20T08:34:43' /YYYY-MM-DDThh:mm:ss observation start, UT
EXPTIME = 0.00000000000000000 /Exposure time in seconds
EXPOSURE= 0.00000000000000000 /Exposure time in seconds
SET-TEMP= -30.000000000000000 /CCD temperature setpoint in C
CCD-TEMP= -30.000000000000000 /CCD temperature at start of exposure in C
XPIXSZ = 9.0000000000000000 /Pixel Width in microns (after binning)
YPIXSZ = 9.0000000000000000 /Pixel Height in microns (after binning)
XBINNING= 1 /Binning factor in width
YBINNING= 1 /Binning factor in height
XORGSUBF= 0 /Subframe X position in binned pixels
YORGSUBF= 0 /Subframe Y position in binned pixels
READOUTM= '1 MHz ' / Readout mode of image
IMAGETYP= 'Bias Frame' / Type of image
FOCALLEN= 1877.0000000000000 /Focal length of telescope in mm
APTDIA = 500.00000000000000 /Aperture diameter of telescope in mm
APTAREA = 178678.08714509010 /Aperture area of telescope in mm^2
SBSTDVER= 'SBFITSEXT Version 1.0' /Version of SBFITSEXT standard in effect
SWCREATE= 'MaxIm DL Version 6.09 160704 03UA3' /Name of software
SWSERIAL= '03UA3-CEEN7-J299Y-94VNY-U13XJ-E0' /Software serial number
SITELAT = '-30 28 15' / Latitude of the imaging location
SITELONG= '-70 45 54' / Longitude of the imaging location
JD = 2458868.8574421294 /Julian Date at start of exposure
OBJECT = ' '
TELESCOP= ' ' / telescope used to acquire this image
INSTRUME= 'FLI ' / instrument or camera used
OBSERVER= ' '
NOTES = ' '
FLIPSTAT= ' '
OBJCTRA = '11 41 57.8' / Nominal Right Ascension of center of image
OBJCTDEC= '-00 00 00.2' / Nominal Declination of center of image
CENTAZ = '240.8503' / Nominal Azimuth of center of image in deg
CENTALT = '55.4947 ' / Nominal Altitude of center of image in deg
HISTORY Automated by CCD Commander
CSTRETCH= 'Medium ' / Initial display stretch mode
CBLACK = 995 /Initial display black level in ADUs
CWHITE = 1011 /Initial display white level in ADUs
PEDESTAL= 0 /Correction to add for zero-based ADU
SWOWNER = 'Ivan Rubtsov' / Licensed owner of software
Vemos que en este caso tenemos la clave IMAGETYP donde el valor de dicha clave nos indicará el tipo de archivo que tenemos.
[9]:
bias_header[0]['IMAGETYP']
[9]:
'Bias Frame'
Inspección de los bias¶
A continuación vamos a inspeccionar los datos de nuestros bias para comprobar que estos sean homogéneos.
Para ello vamos a generar tres listas para guardar los datos de la mediana, media y la desviación estándar. Estas listas nos servirán posteriormente para analizar dichos datos.
Para hacer una inspección rápida vamos a usar la función imstats (definida al principio del notebook).
[10]:
median_bias = []
mean_bias = []
std_bias = []
for bias in range(len(bias_list)):
median_bias.append(np.nanmedian(bias_data[bias]))
mean_bias.append(np.nanmean(bias_data[bias]))
std_bias.append(np.nanstd(bias_data[bias]))
print(imstats(bias_data[bias]))
{'npix': 16777216, 'min': 956, 'max': 3397, 'mean': 996.7283105254173, 'median': 997.0, 'std': 7.704407482434606}
{'npix': 16777216, 'min': 956, 'max': 2297, 'mean': 997.306103348732, 'median': 997.0, 'std': 7.725900408644118}
{'npix': 16777216, 'min': 956, 'max': 2070, 'mean': 997.6139938831329, 'median': 997.0, 'std': 7.718305477278788}
{'npix': 16777216, 'min': 959, 'max': 2539, 'mean': 997.5960484147072, 'median': 997.0, 'std': 7.745906291558662}
{'npix': 16777216, 'min': 960, 'max': 1779, 'mean': 997.6821790933609, 'median': 998.0, 'std': 7.7119550845261315}
{'npix': 16777216, 'min': 958, 'max': 3624, 'mean': 997.5545263886452, 'median': 997.0, 'std': 7.778426647065897}
{'npix': 16777216, 'min': 952, 'max': 4703, 'mean': 997.694066464901, 'median': 998.0, 'std': 7.777878386898218}
{'npix': 16777216, 'min': 957, 'max': 3065, 'mean': 997.551256775856, 'median': 997.0, 'std': 7.733513161549659}
{'npix': 16777216, 'min': 954, 'max': 1640, 'mean': 997.4676412343979, 'median': 997.0, 'std': 7.698876510243282}
{'npix': 16777216, 'min': 954, 'max': 1463, 'mean': 997.5170595645905, 'median': 997.0, 'std': 7.688641429875919}
{'npix': 16777216, 'min': 957, 'max': 65535, 'mean': 997.463648557663, 'median': 997.0, 'std': 26.597340537671517}
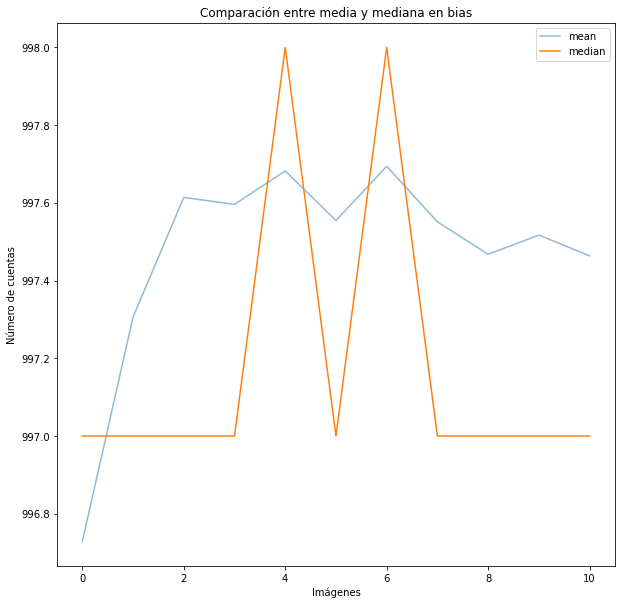
Graficamos la media y mediana¶
Comparamos la media y la mediana de los valores de cada pixel de los bias. Aquí podremos ver que la mediana nos da valores más estables que la media simplemente por la definición de estas funciones.
[11]:
plt.figure("Bias", figsize=[10, 10])
plt.plot(np.arange(0,len(bias_list)),mean_bias, alpha=0.5, label='mean')
plt.plot(np.arange(0,len(bias_list)), median_bias, label='median')
plt.title('Comparación entre media y mediana en bias')
plt.xlabel('Imágenes')
plt.ylabel('Número de cuentas')
plt.legend()
#plt.imshow(stacked, vmin=1000, vmax=2000, origin='lower')
#plt.colorbar(label='Counts')
plt.show()
Graficamos la desviación estándar¶
Al igual que con la media y la mediana vamos a representar la desviación estándar de los bias. La desviación estándar nos servirá para eliminar bias que tengan unos valores muy dispares en diferentes pixeles de la imagen, ya porque haya entrado luz a la hora de tomar estos bias o por lectura del sensor.
[12]:
%matplotlib inline
plt.figure("Bias", figsize=[10, 10])
plt.plot(np.arange(0,len(bias_list)),std_bias, alpha=0.5, label='std')
plt.title('Desviacián estandar')
plt.xlabel('Imágenes')
plt.ylabel('Número de cuentas')
plt.legend()
#plt.imshow(stacked, vmin=1000, vmax=2000, origin='lower')
#plt.colorbar(label='Counts')
plt.show()

Eliminación de los bias «extraños»¶
Hemos comprobado que hay bias que tienen valores en pixeles muy dispares. Vamos a eliminar aquellos que se encuentren por encima de un tanto por ciento del valor de la desviación estándar. En este caso eliminaremos los que tengan una desviación estándar mayor que un 20% (en total se elimina una imagen):
[13]:
for r in range(len(bias_list)):
if std_bias[r] > np.median(std_bias)*1.2:
print(bias_list[r])
bias_list.remove(bias_list[r])
imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0547_000001089.fit
Por tanto en bias_list tendremos nuestra nueva lista de bias, que serán los que usaremos para realizar el superbias.
[14]:
bias_list
[14]:
['imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0534_000001060.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0535_000001061.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0535_000001062.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0536_000001063.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0536_000001064.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0536_000001065.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0537_000001066.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0537_000001067.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0538_000001068.fit',
'imagenes/calibracionImagenes/bias/Telescope3_BIAS_1x1_0s_-30degC_20200120_0538_000001069.fit']
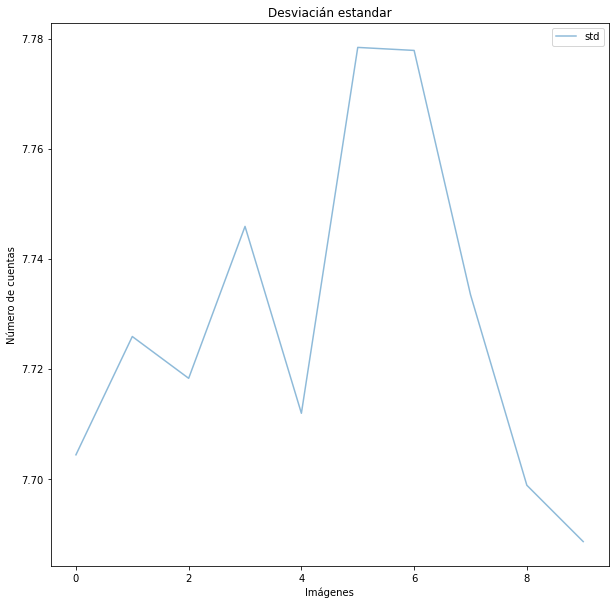
Graficamos de nuevo la desviación estándar¶
Aquí ya vemos la desviación estándar de cada bias, vemos que hay variaciones pequeñas por lo tanto los tendremos listos para generar el superbias.
[15]:
plt.figure("Bias", figsize=[10, 10])
plt.plot(np.arange(0,len(bias_list)),std_bias[:-1], alpha=0.5, label='std')
plt.title('Desviacián estandar')
plt.xlabel('Imágenes')
plt.ylabel('Número de cuentas')
plt.legend()
#plt.imshow(stacked, vmin=1000, vmax=2000, origin='lower')
#plt.colorbar(label='Counts')
plt.show()
Creación del superbias¶
El superbias no es más que realizar una media o mediana de cada pixel en cada imagen. A este proceso se denomina stacking.
Generamos una nueva lista donde se van a encontrar nuestros datos para realizar el stacking. Para ello abrimos los datos de los bias y se lo asignamos a esta nueva lista:
[16]:
superBias = np.nanmedian(bias_data, axis=0)
bias_header[0]['history'] = f"Esta imagen es un stacking de {len(bias_list)} imagenes"
[17]:
bias_header[0]['history']
[17]:
Automated by CCD Commander
Esta imagen es un stacking de 10 imagenes
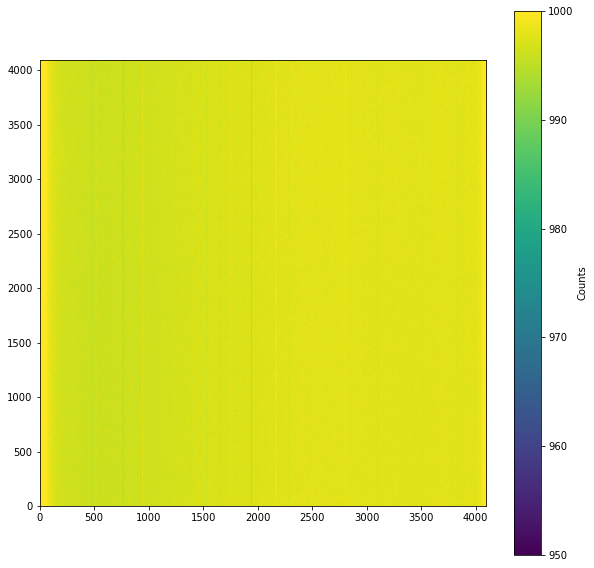
Comprobamos el superbias¶
Una vez que hemos generado el superbias podemos comprobarlo tanto estadísticamente como gráficamente.
[18]:
imstats(superBias)
[18]:
{'npix': 16777216,
'min': 972.0,
'max': 1496.0,
'mean': 997.3878480195999,
'median': 997.0,
'std': 3.2273289419778033}
[19]:
plt.figure("SuperBias", figsize=[10, 10])
plt.imshow(superBias, vmin=950, vmax=1000, origin='lower')
plt.colorbar(label='Counts')
plt.show()
Guardado del superbias¶
Finalmente guardamos el superbias que hemos generado como un archivo FITS a través del método .PrimaryHDU()
[20]:
hdu_Bias = fits.PrimaryHDU(data=superBias.astype(np.float32), header=bias_header[0])
hdu_Bias.writeto(directorioCalibracion + "superBias.fit", overwrite=True)